
Rendez-vous international de la Simulation, du HPC, du Big Data et de l'IA
Présent tous les ans à l'Ecole Polytechnique de l'Université Paris Saclay depuis 2005
a été transformé en un évènement 100% Digital,
le Teratec Digital Forum 2020
http://www.teratec.eu/forum/Photos_Videos.html
en raison des circonstances générées par la pandémie du COVID-19 et des décisions prises par les gouvernements des pays
dont sont issus les exposants, les conférenciers et les participants,
Mardi 13 et mercredi 14 octobre 2020
présente un participant digital
https://forum-teratec.vimeet.events/fr/sheet/343351/fr
membre et Vice-Président de

Association Internationale d'Arts Numériques Sciences et Technologies,
membre de l'AFPR-FA (Association Française de Prototypage Rapide et Fabrication Additive)

Patrick Saint-Jean, Maître de Conférence ENSC Université Paris Saclay




Issu
de plus de 50 ans de R&D d'un travail personnel et collectif en
Arts Sciences et Technologies dans de nombreuses équipes :
Après Math Sup et Math Spé B au Lycée Lavoisier à Paris, s'orientant en chimie macromoléculaire
(il avait déjà créé son Labo de chimie des Matières Plastiques dans la salle de bain du HLM de ses parents)
mais face aux crises du pétrole qui ne cesseront pas depuis 1967,
il se réoriente vers l'informatique, stimulé par un article de Minsky (MIT)
et le constat d'un plaisir réel à réaliser son premier programme en 1967 (Gamma 40 de la SEP-ESA, Tour Nobel)
Il étudie alors l'Informatique-Electronique-Automatique à l'E.S.I.E.A
où pratiquera les calculateurs analogiques et dès 1972 la mini-informatique (H316, 16 k Honeywell-Bull, IBM 1010),
pour optimiser les mélanges d'essence pour une rafinerie de l'Etang de Berre, les Réseaux à destruction partielle pour la Thomsom
et la répartition des terrains habitables citadins suspendus en structure métalique en Architecture,
où il appliquera ses premières recherches et inventions (trans-combinatoire, texture prétopologique et processus prétopologiques markovien.
Il deviendra Ingénieur (promo 75),
Attaché de Recherche CNRS au CNET-CEMAMu (Conférences et Recherche Musicale qui l'orientera vers invention de l'UPIC V0 pour Iannis Xenakis, 1974-77)
et au CEA IPSN, CEN-FAR, Département de Protection (Caryotype automatique, Docteur LE-GO, 1974-78,
et l'analyse d'image cellulaire pour la pathologie B et T du sytème immunitaire avec Victoria Von Hagen, Biologiste, 1975-86).
Crée les premières SCOP pour la Recherche et le Développement Industriel Economique et Sociale
par les moyens de l'Informatique Electronique Automatique
ISTEMI-SUN (1979-92) et COMTEC (1980) pour la Communication.
sa formation passée en dessin industriel l'aide à déposer des brevets en France et aux USA.
Initie l'Art Holographique à l'UNESCO (1984) avec une Collection du Trésor de la Vaisselle de Trace
réalisée en partenariat avec l'Académie des Sciences de Bulgarie
où il travaillera sur les puces électro-optiques holographiques et convetisseur N/A et A/N neuronaux.
Informatise le Théatre National de Chaillot pour Jérôme Savari, et la Comédie Française pour Antoine Vitez
Forme les intermitants du spectacle et les comédiens à l'informatique (programation, musique, gestion, CEP Université Paris I)
Informatise la gestion de l'UFR d'Arts Plastiques et Sciences l'Art de l'Université Panthéon Sorbonne,
où il devient membre de la Commission Informatique des Universités.
Attaché de Recherche CNRS, Spécialité Génie Biologique et Médical de l'Université de Paris XIII,
Initie La Robotique de Laboratoire (1982) pour la Culture Cellulaire de gliales transformées (Paris XIII Bobigny, 1981-86)
et initie l'Homme Neuro-Glial
et l'Analyse d'Image in vitro dont Perkin's Elmer fabriquera une Hote à flux laminaire Robotisée,
et deviendra Docteur ès-Sciences en Biologie du Développement
Initie les Capteurs de Virus dans un projet sur le système immunitaire et le HIV (Second au Concours INSERM 1989)
Initie la Classification Multihiérarchique et Multiparamétrique
par l'imagerie couleur des mesures multiparamétriques et les textures prétopologiques
(Silvius Laboratoria, Leiden, Holande, 1984-86).
Attaché de Cours en Informatiques et Arts Numériques à l'Université Panthéon-Sorbonne, UFR d'Arts Plastiques et Sciences de l'Art (1974-1996),
Maître de Conférence de l'Ecole Normale Supérieure de Cachan intégrée à l'Université Paris Saclay
(1ère en Mathématiques et 14e en général au Classement international de Shanghai),
Initie le Digital Design, Design Numérique, Nouvelles Technologies et Innovations
au Département d'Arts Appliqués et Création Industrielle, devenu Département Design.
Organise des Laboratoires : LIASA Laboratoire Informatique d'Arts et Sciences de l'Art (UFR APSA, Université Paris I Panthéon Sorbonne),
et LTNIS Laboratoire des Nouvelles Technologies de l'Image et du Son, CREACI Centre de Recheche et d'Etude en Arts et Création Industrielle (ENS-Cachan)
et participe au Centre de Recherche en Psychophysiologie de l'Image et CREIAV Centre de Recherche Expérimental et Informatique des Arts visuels
Patrick Saint-Jean a été Chef de Projet européen pour le réseau EDISNetwork de PolyAgogic CyberSpaces (1996-2000).
Deux fois Excellence Européenne (Medici Italie, Mac Luhan Hollande), et reçoit l'Etoile d'OR 2010 des Voeux de l'Internet.
et continue ses R&D en OPEN-SPACE CREDACI UVI-UCVI,
Centre de Recheche et d'Etude en Design Arts et Création Industrielle,
Université Virtuelle Interactive, Univers Citées Virtuelles Interactives,
pour accueilir des étudiants (BTS, Ingénieurs, artistes, Designers) et coopérateurs (Ars Mathématica, associations, etc.)
Past Vice-President of ACM SIGGRAPH France and Past President d'ACM SIGGRAPH Paris Professional and Student Chapter, Vice-Président d'Ars Mathématica International
Membre de l'AFCET, du CA de l'ASTI, de l'AFIA, de SMF et IHP, d'ACM-SIGAI et d'ACM SIG-HPC, de MaMuPhi ENS-ULM et IRCAM,
Après Math Sup et Math Spé B au Lycée Lavoisier à Paris, s'orientant en chimie macromoléculaire
(il avait déjà créé son Labo de chimie des Matières Plastiques dans la salle de bain du HLM de ses parents)
mais face aux crises du pétrole qui ne cesseront pas depuis 1967,
il se réoriente vers l'informatique, stimulé par un article de Minsky (MIT)
et le constat d'un plaisir réel à réaliser son premier programme en 1967 (Gamma 40 de la SEP-ESA, Tour Nobel)
Il étudie alors l'Informatique-Electronique-Automatique à l'E.S.I.E.A
où pratiquera les calculateurs analogiques et dès 1972 la mini-informatique (H316, 16 k Honeywell-Bull, IBM 1010),
pour optimiser les mélanges d'essence pour une rafinerie de l'Etang de Berre, les Réseaux à destruction partielle pour la Thomsom
et la répartition des terrains habitables citadins suspendus en structure métalique en Architecture,
où il appliquera ses premières recherches et inventions (trans-combinatoire, texture prétopologique et processus prétopologiques markovien.
Il deviendra Ingénieur (promo 75),
Attaché de Recherche CNRS au CNET-CEMAMu (Conférences et Recherche Musicale qui l'orientera vers invention de l'UPIC V0 pour Iannis Xenakis, 1974-77)
et au CEA IPSN, CEN-FAR, Département de Protection (Caryotype automatique, Docteur LE-GO, 1974-78,
et l'analyse d'image cellulaire pour la pathologie B et T du sytème immunitaire avec Victoria Von Hagen, Biologiste, 1975-86).
Crée les premières SCOP pour la Recherche et le Développement Industriel Economique et Sociale
par les moyens de l'Informatique Electronique Automatique
ISTEMI-SUN (1979-92) et COMTEC (1980) pour la Communication.
sa formation passée en dessin industriel l'aide à déposer des brevets en France et aux USA.
Initie l'Art Holographique à l'UNESCO (1984) avec une Collection du Trésor de la Vaisselle de Trace
réalisée en partenariat avec l'Académie des Sciences de Bulgarie
où il travaillera sur les puces électro-optiques holographiques et convetisseur N/A et A/N neuronaux.
Informatise le Théatre National de Chaillot pour Jérôme Savari, et la Comédie Française pour Antoine Vitez
Forme les intermitants du spectacle et les comédiens à l'informatique (programation, musique, gestion, CEP Université Paris I)
Informatise la gestion de l'UFR d'Arts Plastiques et Sciences l'Art de l'Université Panthéon Sorbonne,
où il devient membre de la Commission Informatique des Universités.
Attaché de Recherche CNRS, Spécialité Génie Biologique et Médical de l'Université de Paris XIII,
Initie La Robotique de Laboratoire (1982) pour la Culture Cellulaire de gliales transformées (Paris XIII Bobigny, 1981-86)
et initie l'Homme Neuro-Glial
et l'Analyse d'Image in vitro dont Perkin's Elmer fabriquera une Hote à flux laminaire Robotisée,
et deviendra Docteur ès-Sciences en Biologie du Développement
Initie les Capteurs de Virus dans un projet sur le système immunitaire et le HIV (Second au Concours INSERM 1989)
Initie la Classification Multihiérarchique et Multiparamétrique
par l'imagerie couleur des mesures multiparamétriques et les textures prétopologiques
(Silvius Laboratoria, Leiden, Holande, 1984-86).
Attaché de Cours en Informatiques et Arts Numériques à l'Université Panthéon-Sorbonne, UFR d'Arts Plastiques et Sciences de l'Art (1974-1996),
Maître de Conférence de l'Ecole Normale Supérieure de Cachan intégrée à l'Université Paris Saclay
(1ère en Mathématiques et 14e en général au Classement international de Shanghai),
Initie le Digital Design, Design Numérique, Nouvelles Technologies et Innovations
au Département d'Arts Appliqués et Création Industrielle, devenu Département Design.
Organise des Laboratoires : LIASA Laboratoire Informatique d'Arts et Sciences de l'Art (UFR APSA, Université Paris I Panthéon Sorbonne),
et LTNIS Laboratoire des Nouvelles Technologies de l'Image et du Son, CREACI Centre de Recheche et d'Etude en Arts et Création Industrielle (ENS-Cachan)
et participe au Centre de Recherche en Psychophysiologie de l'Image et CREIAV Centre de Recherche Expérimental et Informatique des Arts visuels
Patrick Saint-Jean a été Chef de Projet européen pour le réseau EDISNetwork de PolyAgogic CyberSpaces (1996-2000).
Deux fois Excellence Européenne (Medici Italie, Mac Luhan Hollande), et reçoit l'Etoile d'OR 2010 des Voeux de l'Internet.
et continue ses R&D en OPEN-SPACE CREDACI UVI-UCVI,
Centre de Recheche et d'Etude en Design Arts et Création Industrielle,
Université Virtuelle Interactive, Univers Citées Virtuelles Interactives,
pour accueilir des étudiants (BTS, Ingénieurs, artistes, Designers) et coopérateurs (Ars Mathématica, associations, etc.)
Past Vice-President of ACM SIGGRAPH France and Past President d'ACM SIGGRAPH Paris Professional and Student Chapter, Vice-Président d'Ars Mathématica International
Membre de l'AFCET, du CA de l'ASTI, de l'AFIA, de SMF et IHP, d'ACM-SIGAI et d'ACM SIG-HPC, de MaMuPhi ENS-ULM et IRCAM,

Le C.R.E.D.A.C.I. est un Think-Tank Opérationnel (TTO)
dans la mesure où le processus de pensée, de réflexion et d'expression voire de poïétique est systémique,
basé sur la Simulation interactive en espaces virtuels à immersion,
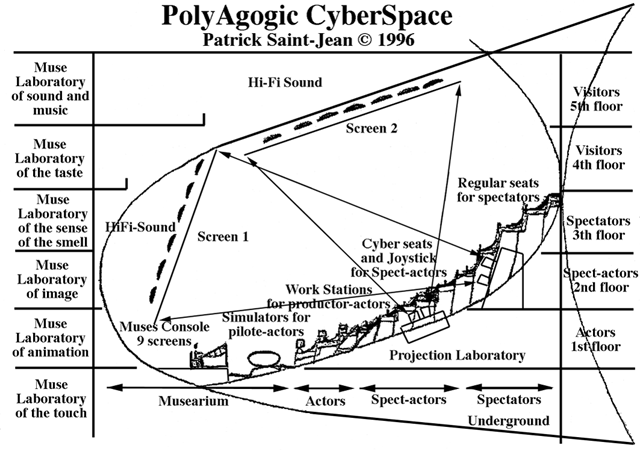
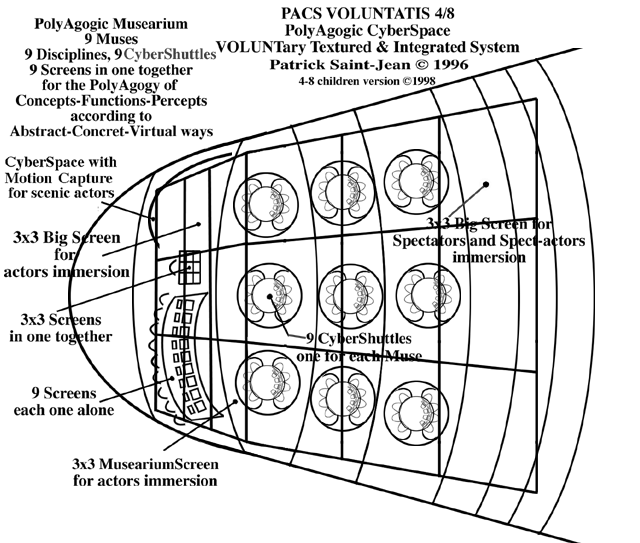
avec le Spectacle de la Connaissance en PolyAgogic CyberSpace composé :
localement en Multi-écrans interactifs et casque de Réalité Virtuelle ou Réalité Augmentée
avec Visio-Conférence, Visio-brieffing-débriffing, Visio-Hackathon, Visio-Workshop et Visio-Lab.
et Simulateur de vol dans les bases de données dynamiques et autostructurantes,
Intelligence Artificielle et Intelligence Personnelle et Collective, Collaborative et Coopérative,
et globalement, dans un Amphithéâtre Interactif à Immersion en réseau,
le tout soutenu par le High Parallel Computing (voire le Huge Optical Parallel Computing)
avec Intelligence Artificielle et DeepLearning Trans-Combinatoire
à Texturologie Quantique Relationnelle à Prétopologie et Topologie Augmentée
dans des Systèmes de Quinternions associés aux TQuBits (QuBits Texturologiques)
formant une Théorie des Sous-Ensembles Intriqués.
http://patrick.saintjean.free.fr/Teratec2020.html
http://patrick.saintjean.free.fr/
http://patrick.saintjean.free.fr/ChoixBibliographiquePSJ.html
Projets du C.R.E.D.A.C.I.
PolyAgogic CyberSpace local :
Virtual OPENSPACE au premier étage
Finition d'architecture intérieure et bureau-d'étude sous verrière avec intallation multi-écrans, projection,
Studio Virtuel Interactif en Réseau de Génie Biologique et Médical sur le Génôme en 3D interactif animatique
et simulation de processus biogénômiques et biogénétiques avec Capteurs de Virus (pathologie et stratégie thérapeutique).
Folding@Home à la Française pour Chercheurs isolés ou Groupes de R&D à distance.
Studio Visio-Conférence interactive, inter-créative et inter-créactive
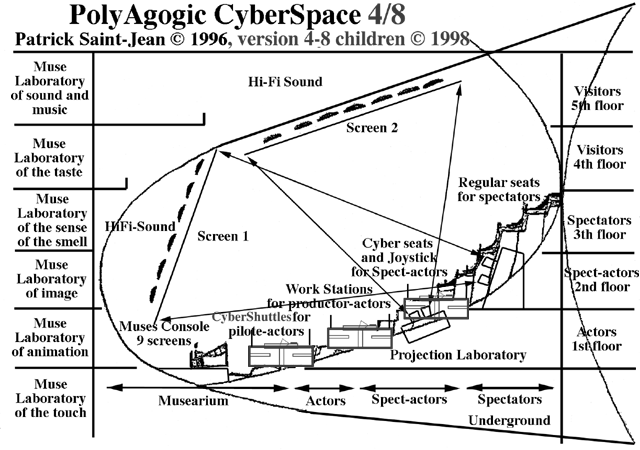
Réalisation de la Version V3.0 du PolyAgogic CyberSpace,
intégrant un multi-piste plus important (8 au lieu de 4 écrans/projecteurs + 8 pistes sons audio et Midi 2.0),
avec la Visio-Conférence, la visualisation 2D et 3D, l'analyse, le traitement, l'animation,
l'IA et la simulation de processus et la synthèse de la 3D, connectée au Big Data et HPC,
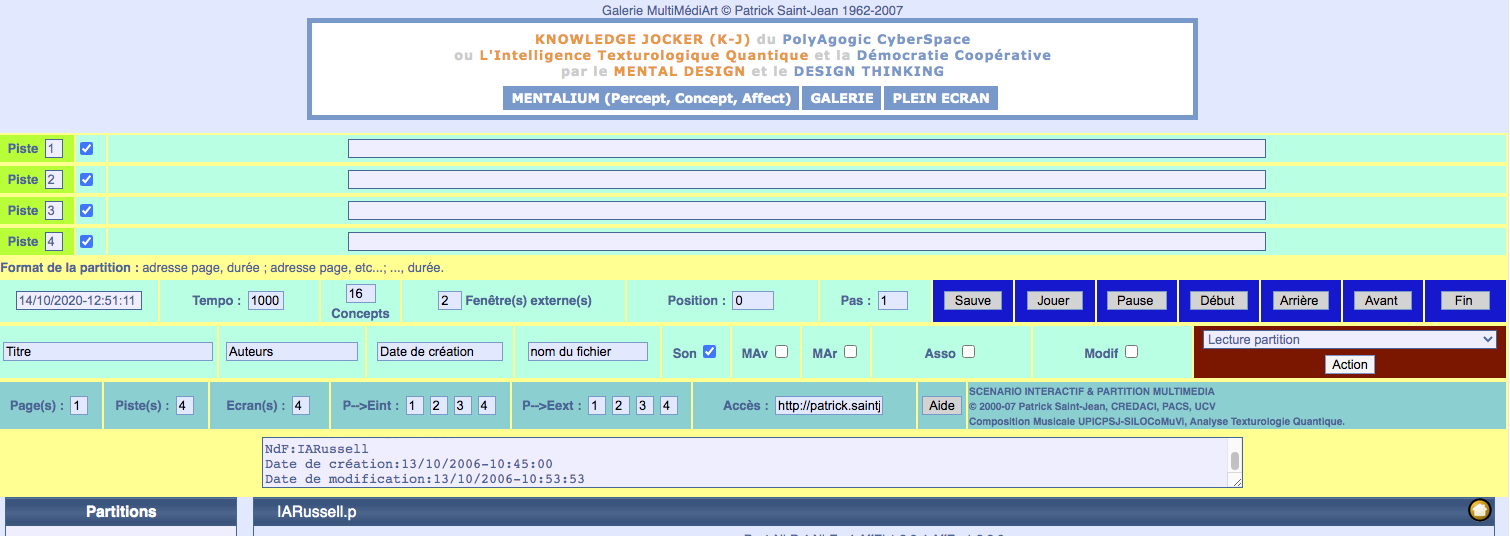
intégrant IanniX, "a graphical open-source sequencer for digital art",
de Thierry Coduys (chef de projet) et Guillaume Jacquemin (développeur), le logiciel de composition musicale
issu de l'UPIC (Unité Polyagogique Informatique du CEMAMu), conçu par Patrick Saint-Jean (sous le nom de SILOCoMuVi, UPIC V0, 1975-77)
pour le Centre d'Etudes de Mathématique et Automatique Musicales 1972, ancien CCMIX 1985, EMAMu 1966 initiés par le Compositeur Iannis Xenakis.

et intégrant le codage musical orchestral Midi 2.0
Organisation de l'OPEN-SPACE
au Rez-de-Chaussée
Bibliothèque Histoire et Philosophie du Numérique et du Vidéo-Numérique
(Computer Sciences, Computer Art, Computer Graphic et Philosophie des Sciences et du Numérique),
OpenSpace Simulation de Vol dans les Espaces de Données Virtuelles,
Galerie d'oeuvres digitales 2D et Vitrines d'Exposition d'oeuvres de 3D Printing
(Titres et Travaux PS-J Arts et Sciences de l'Art)
Espace de Réunion et de Lecture
Espace de projection vidéo lié à la Bibliothèque
Coin Pause et Repas
Ateliers au Rez-de-Jardin
Atelier de 3D Printing, l'impression 3D en art (matériaux, formes et couleurs)
et génie-artistique (enceintes accoutiques)
Banc de montage vidéo-numérique, réalité augmentée et réalité virtuelle,
Salle de Réunion, Zone de réunion en verrière ou extérieure, Espace repos et repas (BBQ).
PolyAgogic CyberSpace Global UVI-UCVI en réseau :
Amphithéâtre Interactif à Immersion pour le Design Numérique Multimédia
pour le Spectacle de la Connaissance avec K-J (Knowledge Jokey)
et l'accès au patrimoine culturel et scientifique,
pour l'éducation et la création artistique multimédia et audiovisuelle en réseau.
Ateliers de formation HPC et Spectacle de la Connaissance.
Ateliers de production pédagogique et bibliothécaire pour alimenter le PolyAgogique CyberSpace
Mise en place de la Version V3.0 du PolyAgogique CyberSpace en réseau
Dans l'Université Virtuelle Interactive transformé en Univers Cités Virtuelles Interactives
où l'Univers est un Poly-Multi-Pluri Diversitaire ( PMPD) relationn.
Recherche Investisseurs pour Construction en face
d'un Centre de Formation HPC-HOPC-UVI-UCVI 2020-2025


Travaux en cours
GBM-LAB
avec FOLDING@HOME In 2020 International Citizen Scientists create an Exascale Computer to Combat COVID-19
https://foldingathome.org/papers-results/
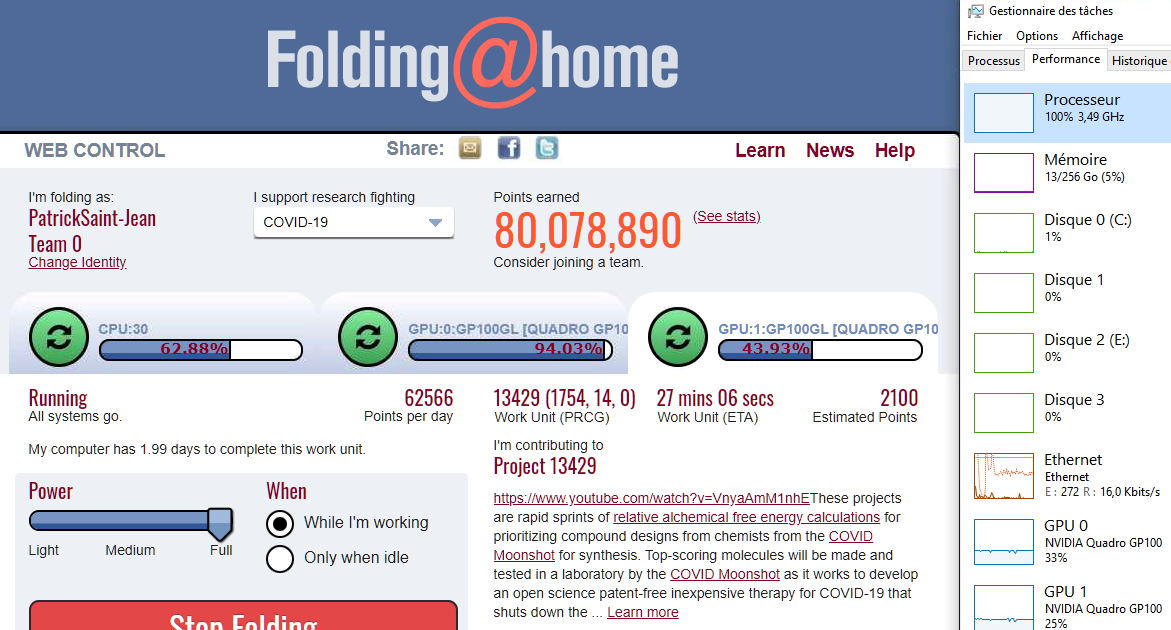
As contributor of Folding@home, Patrick Saint-Jean is folding to support researches fighting COVID-19
with more than 80,000,000 points earned from April to November 2020 and
Contributing to 6 International Groups of Researche :
- Sweden : KTH /SciLifeLab Delemotte Lab
https://www.biophysics.se/index.php/members/sergio-perez-conesa/
https://www.biophysics.se/index.php/projects/delemottelab/
Simulation of a potassium ion channel inactivation and conduction.
Potassium ion channels are crucial proteins of the membranes of excitable cells like neurons or cardiac cells.
They open and close based on stimuli allowing for the conduction of ions (in essence electric impulse) through the membrane.
Their malfunction is related to diseases such as cardiac arrhythmias or epilepsy.
The first COVID19 project from our lab consiste of assembling the envelope protein, which is an ion channel important for viral function.
Learning about how it forms can inform the design of molecules that will prevent proper assembly.
- Washington ity School of Medicine in St. Louis : Greg Bowman's lab
https://bowmanlab.biochem.wustl.edu/software/
Sukrit Singh is a Biophysics PhD student
This project simulates the SARS-Cov2 nsp12 polymerase (RNA-directed RNA polymerase),
which is responsible for duplicating the SARS-Cov2 genome during infection.
These simulations will focus on identifying druggable pockets on the protein's surface as well as predicting the effect of mutations on drug sensitivity.
Artur is a Graduate Student
This project simulates several myosins found in the human body.
Myosins are the proteins responsible for most of the force generated by the body, from the contraction of muscles to the movement of materials around the cell.
Understanding the mechanistic basis of drugs that might be used to treat defects in myosin, including hypertrophic and dilated cardiomyopathies as well as heart failure.
Myosin was featured as the PDB Molecule of the Month in June 2001.
- Temple ity, Department of Chemistry, Philadelphia, Pennsylvania, United States : The Voelz Lab
participates in the Folding@home project, hosting two servers.
Dr. Voelz was formerly a postdoctoral scholar in the Vijay Pande lab at Stanford ity.
http://voelzlab.org
Neha is a postdoc.
Coronavirus SARS-CoV-2 (COVID-19 causing virus) proteins.
These are high-priority projects to simulate the proteins of the COVID-19.
Dylan Novack is PhD student.
https://foldingathome.org/2020/03/10/covid19-update/
https://foldingathome.org/news/
COVID Moonshot designs : https://covid.postera.ai/covid
Fragment HITS from DIAMOND
14676 - FEP screening of 20 fragment hits from DIAMOND UK in solution
14333 - SARS-CoV-2 RBD domain in complex with human neutralizing S230 antibody Fab fragment (PDBs: 6nb8, 2ghv)
http://dx.doi.org/10.1016/j.cell.2018.12.028
SARS-CoV-2 COVID Moonshot absolute free energy calculations generated on
Folding@home now released as an AWS Open Data Set by Vincent Voelz, November 2, 2020
- Hong Kong ity of Science and Technology : Huang Lab
http://compbio.ust.hk/public_html/pmwiki-2.2.8/pmwiki.php?n=Main.HomePage
developing and applying novel computational tools which bridge the gap between experiments and simulations.
Examples of the interested research areas include RNA folding, protein misfolding/aggregration,
conformational changes in Transcription Elongation, and the development of Markov State Models for long timescale dynamics.
a hub for Folding@Home in Asia
- ity of Illinois at Urbana-Champaign : the Shukla Group
https://shuklagroup.org/
to combine theory, computation, and experiments to develop quantitative models of biological phenomena relevant for health, energy, and climate change.
The research program is focused on developing a platform for understanding regulation of protein function
such as elucidating mechanistic insights to regulate plant growth and development in context of global climate change.
This system is the solvated human ACE2 (Angiotension-converting enzyme 2) and the RBD (receptor-binding domain) complex involved in SARS-CoV-2 transfection.
These simulations will allow us to understand the major interactions responsible for binding of these proteins and how the protein behave in the body.
- MSKC, Memorial Sloan Kettering Cancer Center, New York, USA : John Chodera Lab
Computational and Systems Biology Program, Sloan Kettering Institute, https://www.mskcc.org/research/ski/labs/john-chodera
The Projects 17505-08, Disease Type: cancer, are high-temperature vanilla simulation of an apo kinase AURKA : N-MYC complex to explore protein-protein interactions.
https://foldingathome.org/2020/07/28/introducing-covid-moonshot-weekly-sprints-help-us-discover-a-new-therapy/
https://www.youtube.com/watch?v=VnyaAmM1nhEThese projects are rapid sprints of relative alchemical free energy calculations
for prioritizing compound designs from chemists from the COVID Moonshot for synthesis.
Top-scoring molecules will be made and tested in a laboratory by the COVID Moonshot as it works to develop
an open science patent-free inexpensive therapy for COVID-19 that shuts down the essential SARS-CoV-2 main viral protease.
The COVID Moonshot has already made and tested hundreds of compounds, and is pursuing several good lead series.
You can see their progress in real time here: https://covid.postera.ai/covid/submissions/compounds
In addition to helping us prioritize compounds,
you can help purchase more compounds for synthesis at cost from Enamine
by sponsoring the GoFundMe page for patent-free open science COVID-19 drug discovery!
This is a radical new approach to drug discovery that aims to rapidly produce inexpensive new therapies.
This project is managed by at Memorial Sloan Kettering Cancer Center.
http://choderalab.org
The Chodera lab combines expertise in theory, computation, and automated biophysical experiments
to transform physics-based simulations into predictive models
of drug binding, dynamics, and selectivity for the design of anticancer therapeutics.



Atelier de 3D Printing (Impression 3D additive et soustractive)
Covid-19 connected to ACE2 and spike glycoproteine on the capside of covid connected to T4 (3D Printing, Resine AnyCubic),
Visière Z-Glass Zortrax 3D printing (M300, with printing of Covid-Paravent from Geneviève Bonieux)
https://www.arsmathematica.org/IS/index-IS.html
La R&D en Génie Biologique et Médical du CREDACI GBM-LAB reprend les travaux des années 1974-89
(Robotique de Laboratoire pour la culture cellulaire et l'imagerie biomédicale 2D et 3D) de façon plus théorique,
mais avec une volonté de simulation dynamique et interactive 3D des Capteurs de Virus Thérapeutiques
pour compenser la saturation et soulager les systèmes immunitaire et respiratoire,
en assurant le prétraitement des virus après stockage pour être retraités natuellement.
En effet, nous savons que "the air transports viruses and other pathogens.
Since viruses are smaller than other bioaerosols, they have the potential to travel further distances.
In one simulation, a virus and a fungal spore were simultaneously released from the top of a building;
the spore traveled only 150 meters while the virus traveled almost 200,000 horizontal kilometers."
"Aerosols (<5 μm) containing SARS-CoV-1 and SARS-CoV-2 were generated by an atomizer
and fed into a Goldberg drum to create an aerosolized environment.
The inoculum yielded cycle thresholds between 20 and 22, similar to those observed in human upper and lower respiratory tract samples.
SARS-CoV-2 remained viable in aerosols for 3 hours, with a decrease in infection titre similar to SARS-CoV-1.
The half-life of both viruses in aerosols was 1.1 to 1.2 hours on average.
The results suggest that the transmission of both viruses by aerosols is plausible,
as they can remain viable and infectious in suspended aerosols for hours and on surfaces for up to days."
Ainsi faire des capteurs de virus passe par la culture de bioaérosol de type cellulaire.
Travaux en cours
http://patrick.saintjean.free.fr/GBM-LAB.html
Virtual Simulated Thinking LAB
Le principe peut être généralisé à toute base de données :
En effet si un mot était une molécule intégré dans une phrase, un paragraphe, un chapitre, un volume, une collection, une bibliothèque ?
Alors tout mot numérique serait présent dans une multitude d'hypertextes moléculaires différentiés et diversifiés, et interconnectés entre-eux
comme des macromolécules intriquées ayant leur propre structure et fonctionalité.
Comme une protéine, un enzime, ou un virus se déplacent sur les doubles hélices des ADN et ARN,
en utilisant des moteurs prétopologiques d'agrégation et de dispersion, pour se répliquer, se muter, ou interagir sur la structure pour modifier les processus fonctionnels,
les mots, circulant de façon structuro-fonctionnelle, font émerger alors des pensées, fonctions d'onde et scénario d'une simulation d'une conscience environnementale.
Les PolyAgogics CyberSpaces sont faits pour cela à travers le Design Multimédia et le Spectacle de la Connaissance
immersive et auto-intra-inter active, créactive, sensorielle et sensorie-motrice
provoquant la synergie-synectique-synesthésie de l'objet-sujet-projet concret-abstrait-virtuel.
MATH-LAB
A l'aide des Texturologies Quantiques, le High Parallel Computing (HPC) utilisant les Bits-Computers
devient un High Parallel Quantum Computing (HPQC) avec les QBits-Computers
et un Huge Parallel Texturology Quantum Computing (HUPTQC) avec les TQBits-Computers
et un Huge Optical Parallel Texturology Quantum Computing (HUOPTQC) avec les OTQBits-Computers (Optical Texturology QBits)
Avec myQLM d'ATOS,
https://atos.net/en/lp/myqlm
https://myqlm.github.io/index.html
https://myqlm.github.io/myqlm_specific/notebooks.html#
https://mybinder.org/v2/gh/myQLM/myqlm-notebooks/master?filepath=overview.ipynb
nous préparons les NoteBooks myQLM de programmation quantique
https://hub.gke2.mybinder.org/user/myqlm-myqlm-notebooks-8ebi0h95/notebooks/overview.ipynb#
The Atos Quantum Learning Machine (QLM & QLM E) is an enterprise-class solution
for quantum simulation that extends the capabilities of myQLM.
Avec Wolfram Mathematica les NoteBooks sont générés automatiquement à partir du calcul et de la programmation.
As a member of SimTK, a community of biomedical researchers
who share software, models, data, and knowledge and
having a repository of Jupyter notebooks, an open-source web application
that allows you to create and share documents that contain live code, equations, visualizations and narrative text.
Uses include:
data cleaning and transformation, numerical simulation, statistical modeling, data visualization, machine learning,
and With Binder, open those notebooks in an executable environment,
making your code immediately reproducible by anyone, anywhere.
https://gke.mybinder.org/
after enter your repository information by providing in the above form a URL or a GitHub repository
that contains Jupyter notebooks, as well as a branch, tag, or commit hash.
Launch will build your Binder repository.
If you specify a path to a notebook file, the notebook will be opened in your browser after building.
A partir des travaux
sur la Trans-combinatoire et les Textures Prétopologiques et Quantiques et leurs Texturologies
http://patrick.saintjean.free.fr/PACS/Bibliographie/TQ2004/TQ.html
http://patrick.saintjean.free.fr/PACS/Bibliographie/SysOrg89.html
http://patrick.saintjean.free.fr/ASTI-HEBDONo33PatrickSaint-Jean.html
http://ru3.com/luc/tag/people/patrick-saint-jean-cybernetique-intelligence-artificielle.html
ainsi que les Processus Markoviens Prétopologiques
http://patrick.saintjean.free.fr/JourneesdelaPretopologie2011/MoteurSemantiquePretopologiquePACS2011.html
associés à l'Algèbre des Quinternions et à la Théorie des Sous-ensembles superposés et intriqués du Résualisme et de la Cybericité http://acmsiggraphparispc.free.fr/SIGGRAPH2005/HEA2005/HistEtatArtInf.html
http://acmsiggraphparispc.free.fr/SIGGRAPH2005/PSJCACEC.html
http://acmsiggraphparispc.free.fr/CyberFR/CybernetiqueSommaire.html
http://patrick.saintjean.free.fr/SculptureInformatique1993/TechImagesN21DossierFormation1993.html
http://patrick.saintjean.free.fr/PACS/Bibliographie/UPICauPACS/UPICauPACS.html
http://ru3.com/luc/tag/people/patrick-saint-jean-cybernetique-intelligence-artificielle.html
http://patrick.saintjean.free.fr/PretopologieTQTAPSJ.html
http://patrick.saintjean.free.fr/PSJHugeQuantumComputing2018.html
sont préparés, avec Mathematica, des outils d'IA et DeepLearning, classification multi-hierarchique et multi-paramétrique,
avec représentation 2D et 3D interactives en temps réel par DataMining avec simulateur de vol
dans les données en Réalité Virtuelle et Augmentée,
et algorithmique de Trans-Combinatoire (3^(n-1) + 1 possibilités en parallel au lieu de 2^n),
de Textures et Texturologies Quantiques Prétopologiques Relationnelles.
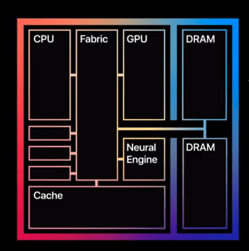
Une R&D avec le processeur M1 d'Apple
(ARM gravure 5 nm, 16 000 000 000 de transistors, 16MG de mémoire cache, 8 CPU et 8 GPU sur une même puce)
devrait aboutir à une écriture des théories et outils également en python.

Ainsi est mis en place tout un processus systémiques de production d'outils et matériels,
de méthodes et démarches, d'interfaces de dialogue et de paramétrage en Design et UX-Design
utilisables en Génie Biologique et Médical en Biochimie cellulaire et macrobiologie génômique.
Ainsi est mise en place tout un processus de production d'outils et matériels,
de méthodes et démarches, d'interfaces de dialogue et de paramétrage en Design et UX-Design.
http://patrick.saintjean.free.fr/MATH-LAB.html
Vous pouvez participer aux R&D ou aux applications de votre choix par e-mail
Recherche de Contrats R&D et de Transferts de Technologies